迄今为止本世纪已经出现了三种冠状病毒的全球流行。2002年11月在中国佛山报道了第一例人类感染严重急性呼吸系统综合症冠状病毒(SARS-CoV)的病例,到2003年7月在全球27个国家中发现了8000多例SARS病例。SARS-CoV感染的主要症状是流感样,包括发烧、头痛、全身乏力、发抖和腹泻。SARS大流行十年后,沙特阿拉伯一名男子被诊断出一种新的冠状病毒,即中东呼吸综合征冠状病毒(MERS-CoV),由于受感染者的国际旅行,MERS-CoV已遍布全球。从2012年9月到2019年12月底,总共报告了2502例经实验室确认的MERS-CoV感染病例,包括858例相关死亡。
在2019年12月,COVID-19爆发,当前的研究表明,这种冠状病毒类似于SARS-CoV。尽管这三种冠状病毒被确定对人类具有高致病性,但尚无有效的抗病毒治疗方法。因此,当前的研究集中在快速开发疫苗和抗病毒药物以预防和治疗新冠状病毒感染。
目前最有吸引力的抗病毒药物靶标之一是SARS-CoV编码的半胱氨酸蛋白酶-木瓜蛋白酶样蛋白酶(PLpro),PLpro参与抑制细胞因子的产生和趋化因子,负责抗病毒感染,所以该酶是SARS-CoV抗病毒药物设计中的重要分子靶标。对CoV-2 PLpro底物特异性、结构和机制的分子了解,将通过进行合理的药物靶向设计和研究来极大地促进有效PLpro抑制剂的研发。
近日《Science》上发表了一篇最新研究,研究人员首先使用新颖的化学方法杂交组合底物库对SARS-CoV-2 PLpro进行了全面的活性分析,结果揭示了控制PLpro底物特异性的分子规则。利用这些信息,接下来设计了具有生化特性的有效抑制剂(VIR250和VIR251),与其他蛋白酶相比,它们对SARS-CoV-2 PLpro和相关SARSCoV-1 PLpro具有高选择性。这项研究工作揭示了控制PLpro底物特异性的分子规律,并为开发具有潜在治疗价值或药物再利用的抑制剂提供了框架。

01
底物特异性概况
在P1位置的甘氨酸基侧的肽键水解会导致nsp1、nsp2和nsp3蛋白的释放,这对于病毒复制至关重要。SARS-CoV-2 PLpro还具有去泛素化和识别这些蛋白质C末端的保守LRGG基序。先前仅针对包含天然氨基酸的组合底物库对SARS-CoV-1 PLpro底物偏好进行的研究,结果表明该蛋白酶识别P4-P1位置的LXGG基序,在P3位置具有广泛的底物特异性。这些结果表明,对结合口袋结构更详细的作图应有助于新的活性底物的设计和用于抑制剂开发工作的最佳肽序列。为了实现这个目标,研究人员开发了一个定义的组合底物库(HyCoSuL),其中包含多种非蛋白原性氨基酸。
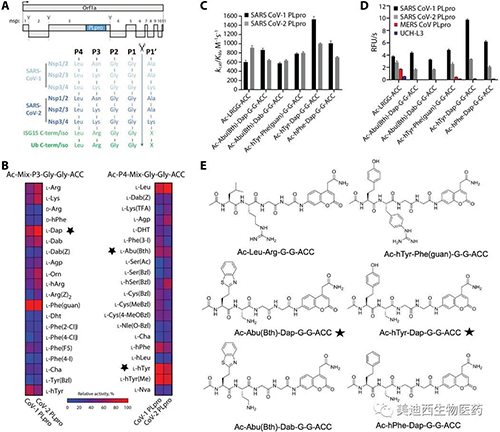
SARS-CoV-1和2 PLpro蛋白酶的活性分析(来源:Science Advances)
由于四肽荧光底物不能被具有去泛素化活性的酶有效地水解,因此研究人员设计并合成了具有Ac-LRXG-ACC和HyCoSuL的一般结构的P2定义文库。位置是固定的,一个位置包含19个氨基酸的等摩尔混合物。通过设计具有针对去泛素酶的定制肽支架的文库,可以在分析过程中达到每个子库中单个荧光底物的最高可能浓度。
P2文库筛选显示,SARS-CoV和SARS-CoV-2 PLpro在此位置具有很高的底物特异性,两种蛋白酶在P3位置均表现出广泛的底物偏好。SARS-CoV和SARS-CoV-2 PLpro的S3口袋不仅可以耐受带正电的残基,而且还可以耐受疏水性氨基酸。这些酶无法识别酸性残基和大多数D-氨基酸。SARS-CoV和SARS-CoV-2 PLpro的S4口袋只能容纳疏水性残基,在天然氨基酸中,几乎只能耐受亮氨酸。SARS-CoV PLpro在P4位置比亮氨酸更好地识别了两个非蛋白残基。
02
PLpro抑制剂的开发
为了进一步分析具有非蛋白原性氨基酸的肽序列的选择性,研究人员通过交换荧光将两种底物转化为抑制剂标记一个反应性基团-乙烯基甲酯(VME)。选择VME组是因为它对DUB具有广泛的反应性,SARS-CoV PLpro和SARS-CoV-2 PLpro抑制剂的动力学分析结果反映了底物水解的结果。与VIR250相比, VIR251对这些酶的抑制作用更强,但选择性更低。两种化合物均显示出对SARS-PLpro变体的高选择性,并能有效抑制SARS-CoV PLpro和SARS-CoV-2 PLpro活性。相反,实际上未观察到对人DUB UCH-L3的抑制,而仅观察到对MERS-PLpro的轻微抑制。此外,将HeLa裂解物与Ub-VME一起温育可产生交联曲线,这种交联曲线不会因滴定VIR250或VIR251而改变。由于已知Ub-VME的主要交联靶标是人DUB酶,因此这些数据表明VIR250和VIR251不会与人DUB发生交叉反应,这是寻找与人DUB具有最小交叉反应性的选择性抗病毒分子的重要发现。
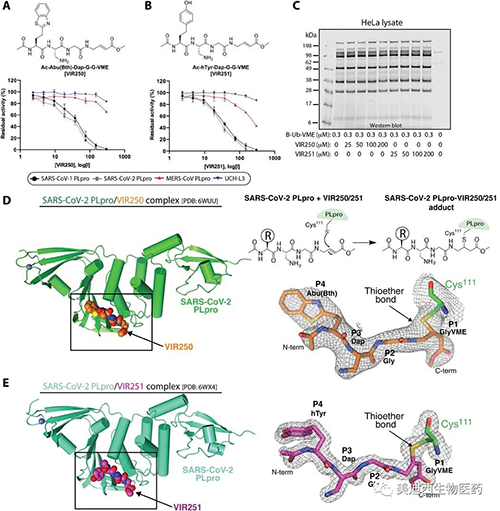
与SARS-CoV-2 PLpro配合使用的非天然氨基酸抑制剂VIR250和VIR251及其晶体结构的表征(来源:Science Advances)
接下来,研究人员着手确定SARS-CoV-2 PLpro与VIR250和VIR251的复合物的晶体结构,以深入了解这些分子抑制SARS-CoV-2 PLpro活性的分子机理,以及观察到的底物选择性的基础。大规模的交联反应产生了CoV-2 PLpro-VIR250和CoV-2 PLpro-VIR251配合物,其产率和纯度足以满足衍射质量晶体的生长。
将载脂蛋白CoV-2 PLpro与CoV-2 PLpro / VIR250和CoV-2 PLpro / VIR251配合物进行比较,发现除β14-β15环位于活性位点附近并经历构象变化外,总体结构相似可能是由于抑制剂的结合。结构分析表明,从CoV-2 PLpro的催化Cys 111侧链投射出广泛的电子密度,其中VIR250和VIR251的所有原子都可以明确地放置在其中。此外,Cys 111之间的共价键VIR250和VIR251都清晰可见。如预期的那样,VIR250和VIR251抑制剂在活性位点附近均占据CoV-2 PLpro的S4-S1口袋并采用类似的结构,P4位置是VIR250和VIR251之间唯一的化学差异区域,在VIR250中为Abu,在VIR251中为h Tyr。
03
VIR250和VIR251的分子识别
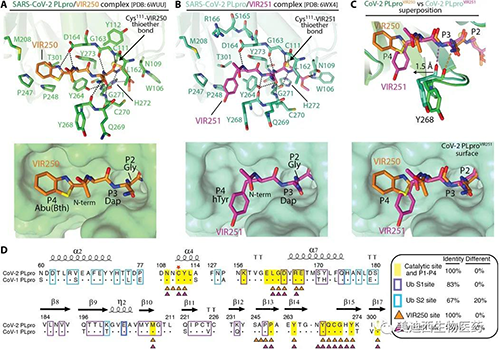
SARS-CoV-2 PLpro对VIR250和VIR251的分子识别(来源:Science Advances)
VIR250和VIR251的P3和P4位置的侧链与CoV-2 PLpro的结合方式有重要区别。与VIR250中的Abu(Bth)相比,P4位置的hTyr向着S4口袋的另一侧突出。β14-β15环(Asn 267、Tyr 268和Gln 269)向VIR251的hTyr转移1.5-Å,促进了这种新的相互作用,从而促进了许多新型的联系,而这些联系在没有这种转变的情况下是无法发生的。值得注意的是,与VIR250和VIR251接触涉及的所有CoV-2 PLpro残基在SARS CoV-1 PLpro中都完全保守,并且两个SARS PLpro变体的总体结构在酶的催化位点非常相似,这可能解释了这些抑制剂具有靶向两种酶的能力。
就上述观察到的底物选择性的相关性而言,P2对Gly的依赖性是来自CoV-2 PLpro的β14-β15和α5-α6环残基的结果。对P3位置的正性和疏水性残基偏爱以及对酸性残基的选择可能是其更宽泛的口袋和靠近Tyr 268、Gln 269和Leu 162的酸性羰基氧的结果。在P4位置上,对庞大的疏水残基的强烈偏爱可以通过P4结合口袋的疏水特性来解释,该特性主要由残基Met 208、Pro 247、Pro 248、Tyr 264和Tyr 268形成。值得注意的是,SARS-CoV-2 PLpro的S4口袋的性质已被VIR250和VIR251的P4处的Abu和h Tyr侧链所利用。
04
CoV-1和CoV-2 PLpro处理Ub和Ubl变体
针对SARS-CoV-1 PLpro进行的研究表明,该酶具有Ub结合域,可有效处理完整的Ub荧光底物,因此研究人员想看看SARS-CoV-2 PLpro是否也是如此。为此使用了基于Ub活性的探针(ABP)来标记这两种酶。研究人员观察到生物素-Ub-VME对两种蛋白酶都进行了实质性标记,其浓度是酶浓度(200 nM)的两倍。但是,与SARS-CoV-2 PLpro相比,SARS-CoV-1 PLpro的标记效率更高。接下来通过测试其与一组Ub / Ubl ABP的反应性来探索SARS-CoV-2 PLpro的Ub和Ubl蛋白底物特异性,该分析的结果表明SARS-CoV-2 PLpro更倾向于识别ISG15和monoUb,并且识别K48连接的和K63连接的diUb的能力较弱。
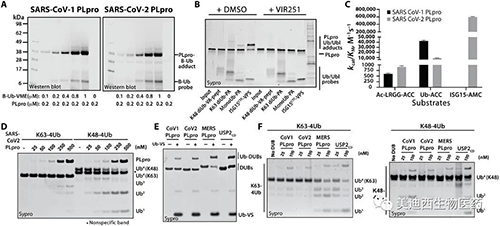
SARS-CoV-1 PLpro和SARS-CoV-2 PLpro对Ub和Ubl变体的处理(来源:Science Advances)
为了更彻底地检查这些差异,研究人员对LRGG-ACC、Ub-ACC和ISG15-AMC荧光底物的SARS-CoV-1和CoV-2 PLpro加工动力学进行了比较。实验结果表明,SARS-CoV-2 PLpro处理Ub-ACC的效率是SARS-CoV-1 PLpro的四倍,并且SARS-CoV-2 PLpro处理ISG15-AMC的效率是Ub-ACC的60倍。此外,SARS-CoV-2 PLpro与SARS-CoV-1 PLpro一样,与K63 tetraUb相比,对K48 tetraUb的处理更为稳健。然而,在并排比较中,与SARS-CoV-1 PLpro相比,SARS-CoV-2 PLpro表现出显著降低了处理K48 tetraUb的能力。
考虑到酶之间的非常高的总体相似性,与SARS-CoV-1 PLpro相比,SARS-CoV-2 PLpro处理K48 polyUb链的能力大大降低了。为了调和这种明显的矛盾,研究人员将SARS-CoV-2 PLpro结构与先前报道的SARS-CoV-1 PLpro结构与K48 diUb配合使用。对SARS-CoV-1和2 PLpro催化位点的比较分析显示,与S1 Ub、VIR250和VIR251的S4-S1位置接触时所涉及的残基100%保守,并且与预期结构非常相似。尽管Ub S1位点比催化位点具有更大的变异性,但总体氨基酸保守性仍然很高,并且结构在该区域排列得很好。
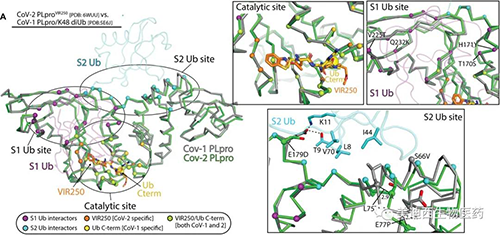
观察到的SARS-CoV-1 PLpro和SARS-CoV-2 PLpro的Ub / Ubl加工概况的分子基础(来源;Science Advances))
与SARS-CoV-1 PLpro的催化位点和S1 Ub位点相比,SARS-CoV-2 PLpro的S2 Ub位点在氨基酸水平上的保守性要低得多,并且在这些区域有一些结构差异对于分子识别S2 Ub很重要。研究人员认为SARS-CoV-2 PLpro处理K48 polyUb的能力下降的主要原因是在S2 Ub结合位点的差异,与此假设一致,SARS-CoV-1 PLpro亮氨酸75突变为丝氨酸导致K48 diUb的结合减少了五倍,而对monoUb没有明显影响。结合SARS-CoV-2 PLpro中的其他变化,SARS-CoV-1和2 PLpro之间在S2结合位点出现了一些相对较小的变化,这些变化对它们处理K48 polyUb的能力的影响很大。
05
结果与讨论
SARS-CoV-2 PLpro是抗病毒药物开发的绝佳候选者,因为它不仅阻断病毒复制,而且还抑制感染细胞中信号级联的失调。通过对药物靶向的合理设计和研究,对SARS-CoV-2 PLpro底物特异性、结构和机制的详细理解将极大地促进有效PLpro抑制剂的开发,这是此项研究的重点。
研究人员利用获得的有关控制SARS-CoV-2 PLpro底物选择性的分子规则的信息来开发共价抑制剂VIR250和VIR251。这些抑制剂被证明具有活性并选择性抑制SARS-CoV-1和-2 PLpro,但对MERS-PLpro的活性却弱得多,而对人DUB UCH-L3的活性则几乎没有。对于进行针对该酶的肽类抗病毒化合物的研究,这是有价值的信息。VIR250和VIR251与SARS-CoV-2 PLpro配合使用的晶体结构揭示了它们的抑制机制,并为观察到的底物特异性谱提供了结构基础。
此外,出乎意料的发现是,VIR250和VIR251的P4氨基酸占据了SARS-CoV-2 PLpro宽S4口袋的相对侧,并且该口袋中还有其他区域未被任一抑制剂所结合,这意味着未来的药物开发工作旨在产生更多有效的抑制剂。对SARS-CoV-2-Mpro和SARS-CoV-2 PLpro底物特异性的比较分析表明,它们具有明显不同的底物特异性,这表明对于肽类抑制剂,不可能设计出一种同时作用于两种酶的抑制剂。但是,如果分别为这两种蛋白酶找到了肽类抑制剂,那么有可能需要将它们用作混合物。另一种可能的方法是寻找可以同时抑制Mpro和PLpro的小分子抑制剂,这种抑制剂在COVID-19的治疗中肯定会非常有益,但它可能与人半胱氨酸蛋白酶发生交叉反应,从而可能导致不良副作用。
结构分析显示,SARS-CoV-2 PLpro的S2 Ub结合位点存在细微的结构和序列变异,研究人员共同认为这会削弱K48 polyUb的S2 Ub结合及随后被加工的能力。此外,对Ub-ACC底物的酶动力学分析表明,该酶可以有效地对其进行处理,但是在SARS-CoV-1 PLpro的情况下,四肽底物和Ub之间的差异仅为10倍左右。这表明两种酶之间在外位结合区相互作用的方面与氨基酸的同一性和相似性有些不同。鉴于Ub和ISG15偶联在逃避宿主先天免疫反应中的作用,SARS-CoV-1和-2 PLpro功能的这些有趣差异是否会对病毒生物学产生任何影响尚待观察,并且将是未来研究的主题。
总的来说,此项研究工作揭示了控制PLpro底物特异性的分子规则,并揭示了底物结合口袋中SARS-CoV-1和-2 PLpro之间非常高的序列和结构相似性。这些发现表明,先前发现的有关SARS-CoV-1 PLpro的信息可立即用于寻找有效的抗病毒分子,并重新靶向抑制SARS-CoV-2 PLpro的已知药物。此外,与SARS-CoV-2 PLpro结合的新型抑制剂VIR250和VIR251的结构为合理开发具有增强效价和ABP的抑制剂提供了框架。此项研究的谱图和晶体学研究为开发性能改善的抑制剂开辟了新途径,研究数据也为设计一种可以同时对抗SARS-CoV-1 PLpro和SARS-CoV-2 PLpro的全选择性抑制剂提供了帮助。美迪西作为新药研发CRO会持续关注此项研究进展。














 相关新闻
相关新闻